
 |
|
|
|
SIS: Système "Susceptible - Infectieux" en épidémiologieB. Bonté, P. BommelUn modèle SIS (Système Susceptible - Infecté) simple, qui permet aux utilisateurs de comparer 3 types de paradigmes sur un même modèle simple en épidémiologie : équations différentielles continues, modèle discret et plusieurs implémentations d'un SMA. ObjectifIl s'agit d'une réplication d'un modèle épidémiologique standard qui simule la propagation d'une épidémie dans une société : des personnes en bonne santé (également appelées Susceptibles) peuvent devenir malades (Infectées) lorsqu'elles entrent en contact avec d'autres personnes déjà porteuses de la maladie. À leur tour, elles contribuent à la propagation de la maladie lorsqu'elles rencontrent d'autres personnes.Dans ce modèle, la maladie n'est pas dangereuse. Au bout d'un certain temps, les personnes infectées redeviennent susceptibles. Une simulation doit donc surveiller le nombre de personnes infectées et de personnes en bonne santé au fil du temps. Ce modèle présente deux versions principales : Version 1: modèle global (équations différentielles)Dans la première variante, la version "globale", les individus ne sont pas représentés, mais seulement les populations de Susceptibles et Infectés : S: nombre d'individus susceptibles. I: nombre d'individus infectés. β: taux d'infection (0,3). γ: taux de récupération (0,1) Ce schéma correspond à un système à 2 équations différentielles : 
Ce modèle continu peut être discrétisé en transformant dt en deltaT : 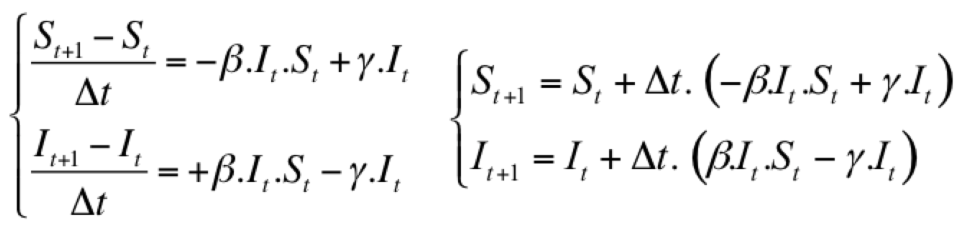 Vue du modèle sous Vensim (modèle "stock & flow") : 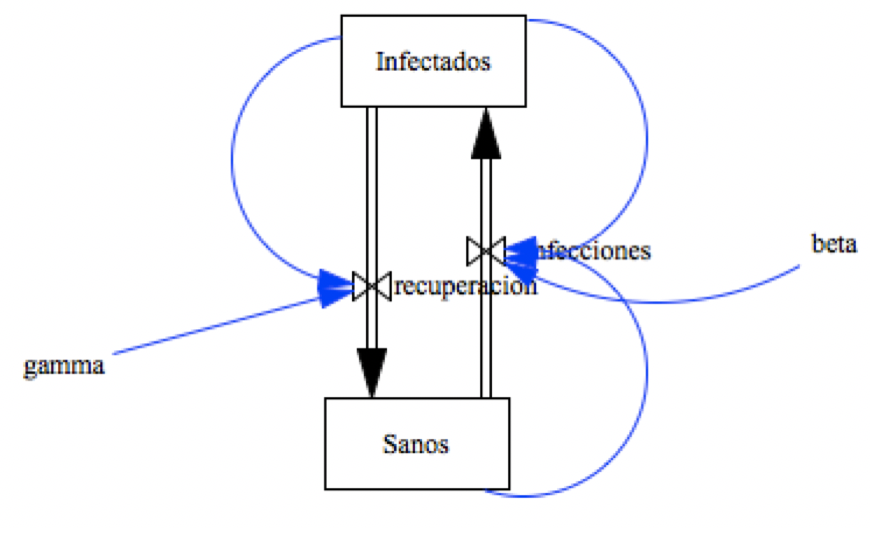 La figure suivante montre des simulations avec Vensim à partir de 3 états initiaux différents : 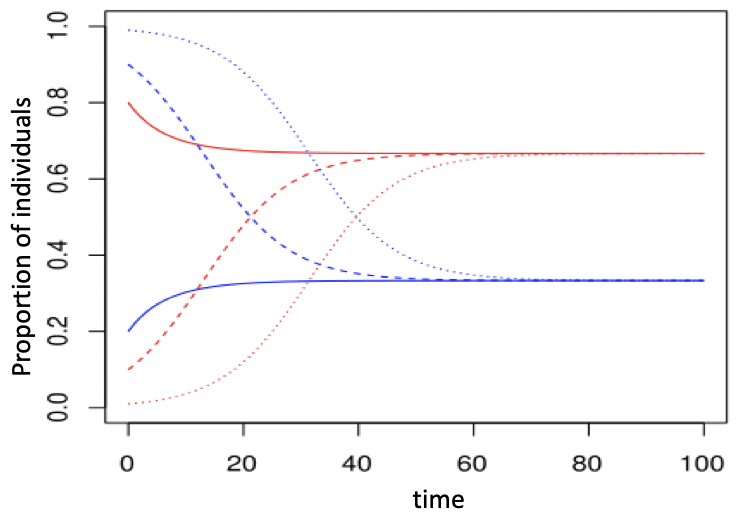 A noter que à l'équilibre : 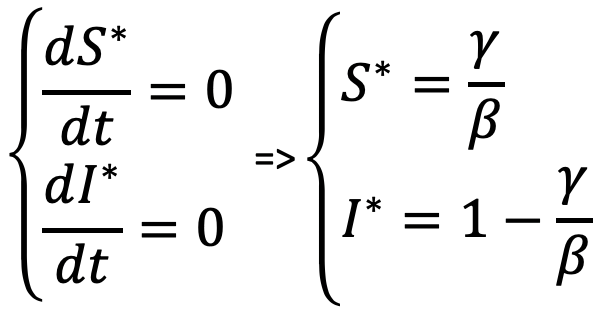 Au bout d'un certain temps, S et I sont stables
; leur niveau dépend uniquement de β et γ : S*=1/3 and I*=2/3
Au bout d'un certain temps, S et I sont stables
; leur niveau dépend uniquement de β et γ : S*=1/3 and I*=2/3
Version 2: ABMLa deuxième variante, "ABM", représente explicitement les individus du système.Le taux d'infection (β = 0,3, défini au niveau populationnel) devient désormais une probabilité individuelle d'être infecté au contact d'un autre agent infecté (30 %). Le taux de guérison doit également être adapté au niveau individuel. Par exemple, il peut être transformé en une Période Infectieuse de 10 jours. Après cette période, l'agent malade se rétablit. En d'autres termes, le taux de guérison (10 %, défini au niveau de la population) se traduit par une période de guérison de 10 jours, définie au niveau individuel. 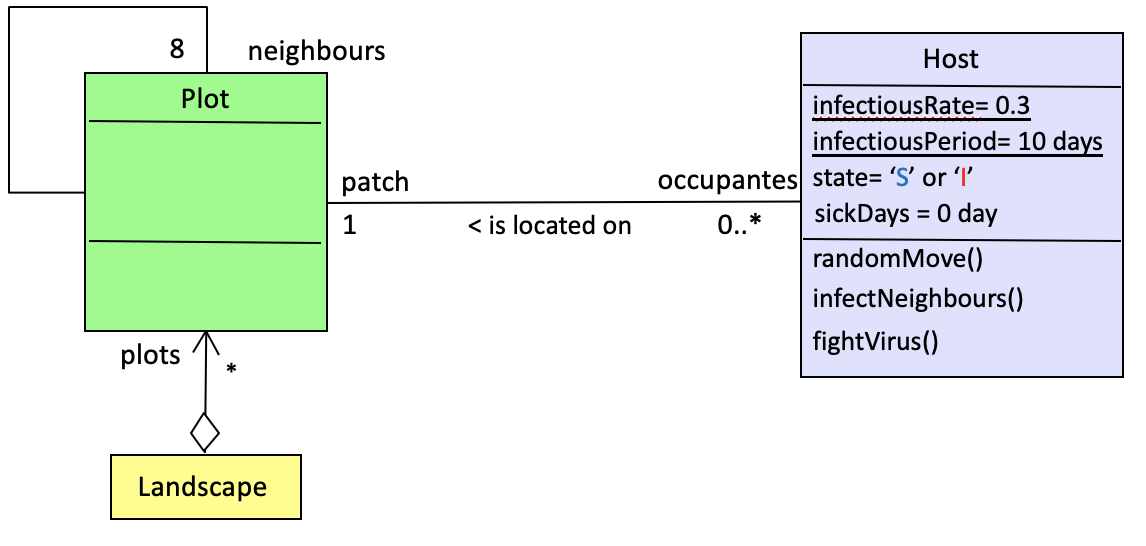 Lorsqu'ils se déplacent dans le paysage, les agents peuvent se rencontrer au hasard. Afin d'avoir une probabilité de rencontre équivalente à celle du modèle global, la taille de l'espace doit être calculée en fonction de la taille de la population. Le paramètre ß (taux d'infection : 0,3) peut être exprimé sous la forme : ß = -k log(1 - c) , où * k est le nombre moyen de contacts entre deux personnes par unité de temps, et * c est la probabilité que le contact entre un individu sensible et un individu infectieux entraîne une infection. Donc c est égal au taux d'infection (= 0,3). En fixant c à 0,3, nous calculons k = -ß / log(1 - c) = -0,3 / log (0,7) = -0,3 / (0,7 log) = 1,93671 Il y a en moyenne 1,93671 contacts entre deux personnes par étape. Ainsi, pour un voisinage de 8 (Moore), 1 agent a 1 chance sur 8 d'aller sur la cellule d'un agent voisin. Si X.Y est la taille de la grille, alors popSize = k * X.Y/ 8 <=> X.Y = popSize * 8 /k Dans Cormas, cette implémentation permet de représenter une grille spatiale avec les agents situés : 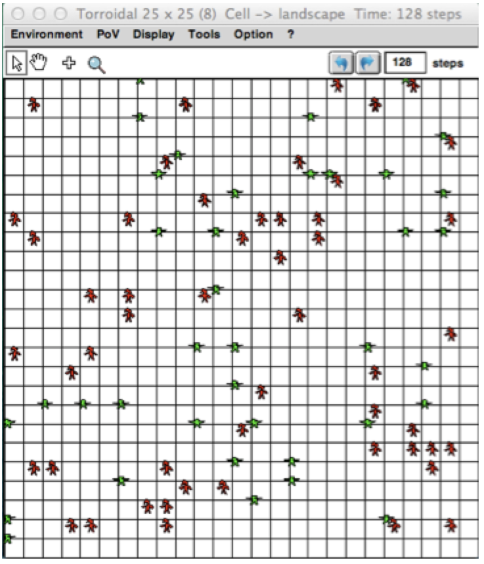 Mais cette transformation du modèle global (éq. différentielle) en un SMA correspond à une façon de faire (le scénario “No Spatial, Infectious Period”). Il peut être défini différemment, comme présenté dans ce qui suit : Implémentions et comparaison des scenarios suivantsEn utilisant le modèle SIS dans Cormas, vous pouvez choisir un scénario parmi 7 :  Pour chaque scénario, les résultats du modèle à équations discrètes (modèle global) et du modèle SMA (modèle local) peuvent être comparés (aggregINFECTED vs indivINFECTED). Ils sont initialisés avec au moins 1000 hôtes. Tous les paramètres sont présentés dans cette table: 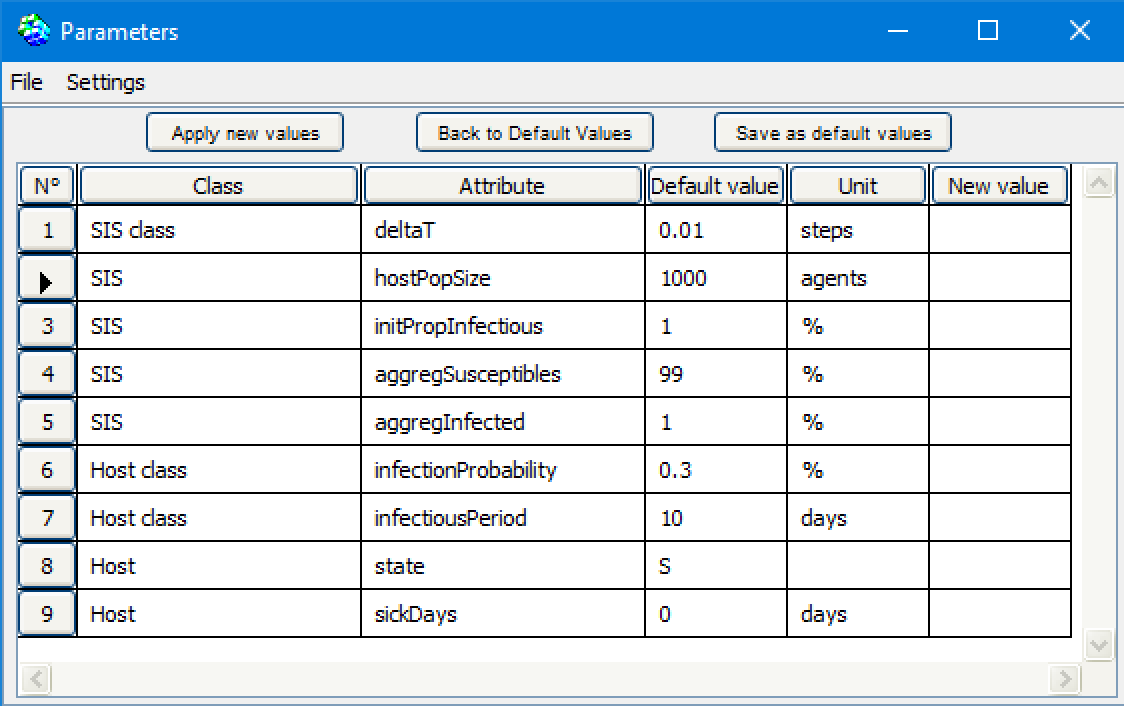 a) macroIci, les agents ne font rien, ou plutôt ne décident de rien. Leur état est modifié par un opérateur extérieur : c'est le résultat du modèle global.Dans cette version (a_macro_deltaT1), le modèle calcule le nombre d'agents infectés et susceptibles selon l'équation discrète avec delta T = 1. Les sorties sont comparées avec celles du modèle global discret avec un deltaT fixé à 0.01 :  Ici, les courbes pour les deux scénarios (agrégés et individuels) sont quasi similaires, ce qui est logique. La petite différence est due à la résolution du temps: deltaT = 1 dans le cas de l'ABM et deltaT = 0.01 pour le modèle global. b) “No Spatial, No Memory”Les agents ne sont pas situés. Ils se rencontrent au hasard. On traduit cela de la façon suivante : chaque agent infecté peut rencontrer un des autres agents pris aléatoirement dans la population. Si ce dernier est Susceptible, alors il peut devenir infecté avec une probabilité donnée (infectionProbability = 0.3).Lorsqu'il est infecté, un agent a une probabilité de 10 % de se rétablir chaque jour. 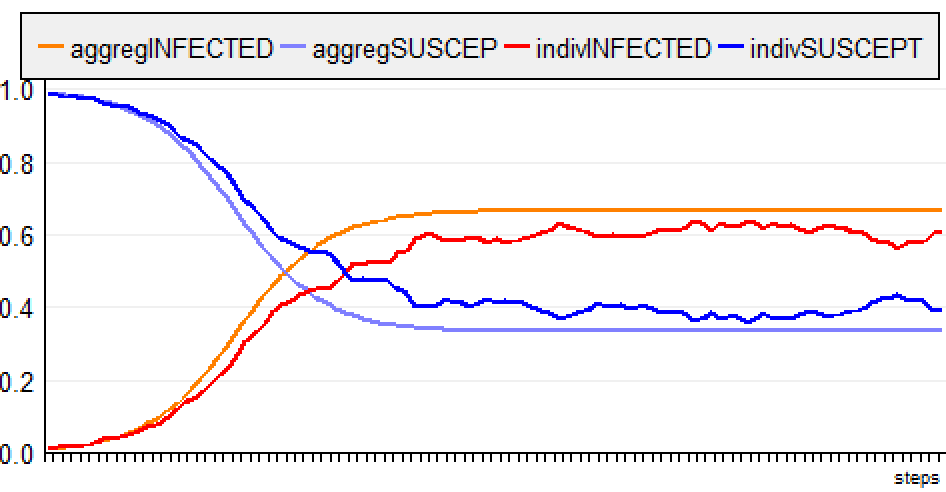 Pour la version ABM, l'évolution de la maladie est un peu plus lente que pour le modèle global et le taux d'infection se stabilise autour 0.6 (au lieu de 0.666). c) “No Spatial, Infectious Period”Les agents ne sont pas situés. Ils se rencontrent au hasard. Lorsqu'il est infecté, un agent est contagieux pendant 10 jours.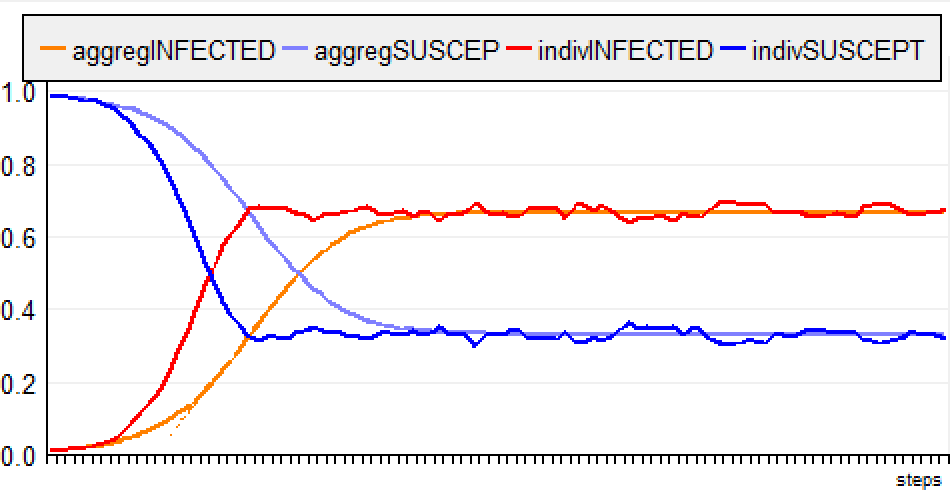 Pour la version ABM, l'évolution de la maladie est plus rapide, mais le taux d'infection se stabilise autour de 0.666 (comme pour le modèle global). d) “NoMemory”Les agents sont situés sur une grille (pour 1000 agents, la dimension spatiale est fixée à 65x64 cellules = 4160 cellules). Ils peuvent se rencontrer lorsqu'ils se déplacent au hasard.Lorsqu'il est infecté, un agent a une probabilité de 10 % de se rétablir chaque jour. Le step général du modèle (et pour les scénarios suivants) est divisé en 3 phases : 1) Tous les agents bougent, 2) Les agents infectés peuvent infectés leurs voisins localisés sur la même cellule, avec infectionProbability), 3) Les agents infectés combattent le virus (probabilité de guérir de 10% à chaque step) 
(la simulation est exécutée sur 150 steps, afin de vérifier que les courbes sont finalement alignées avec l'état d'équilibre). Pour la version ABM, l'évolution de la maladie est bien plus lente, mais mais le taux d'infection se stabilise autour de 0.666 (comme pour le modèle global). e) “Infectious Period”Les agents sont situés et se déplacent de manière aléatoire. Lorsqu'il est infecté, un agent est contagieux pendant 10 jours.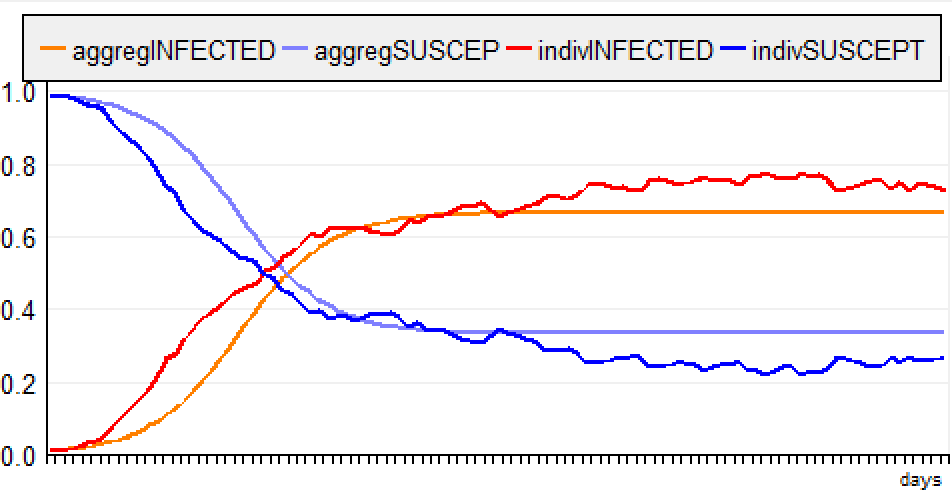 f) “Jump NoMemory”Les agents sont localisés et sautent aléatoirement à un autre endroit de la grille. Lorsqu'il est infecté, un agent a une probabilité de 10 % de se rétablir chaque jour.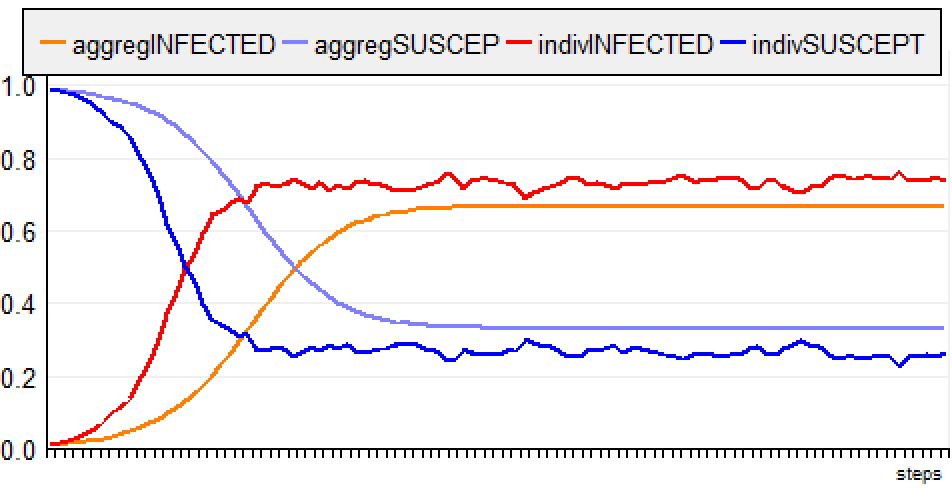
g) “Jump, Infectious Period”Les agents sont situés et sautent aléatoirement à un autre endroit de la grille. Lorsqu'il est infecté, un agent est contagieux pendant 10 jours.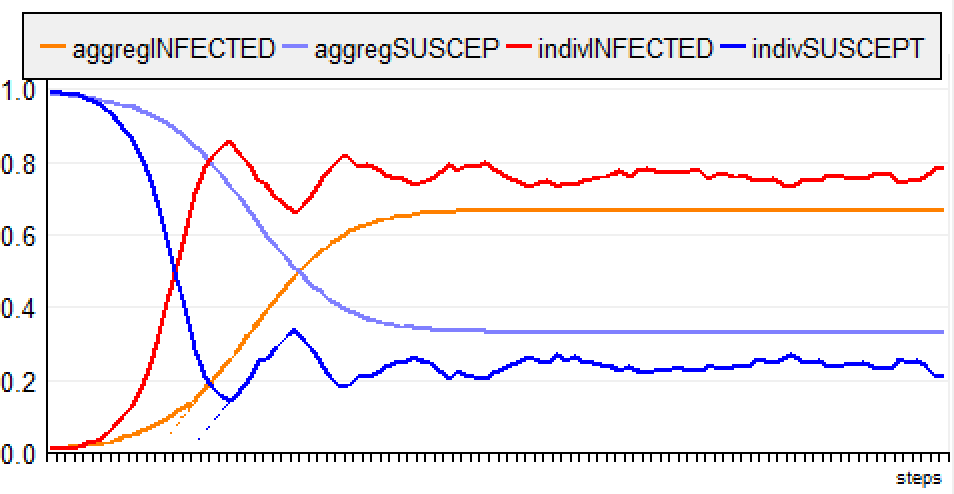 Enfin, la figure suivante permet de comparer un modèle similaire conçu selon 3 types de paradigmes : équation différentielle continue (lignes épaisses), équation différentielle discrète (lignes fines) et ABM-e (lignes pointillées) :  ConclusionMême pour un modèle très simple, il existe plusieurs façons de le formaliser, ce qui conduit souvent à des simulations similaires mais non identiques. Pour les deux modèles "globaux" (équations différentielles continues et discrètes), on constate déjà de petites différences lorsque le delta T = 1 (cf. figure précédente).La traduction de ce modèle en un SMA nécessite des ajustements (pour passer du global au local) avec des conséquences sur les résultats des simulations. Dans le contexte du Covid-19, des initiatives similaires de modélisation didactique ont été menées, avec Gama (https://gama-platform.github.io/covid19) et avec Netlogo (https://covprehension.org/, en français).
|
|